がん組織中のRET融合遺伝子の検出
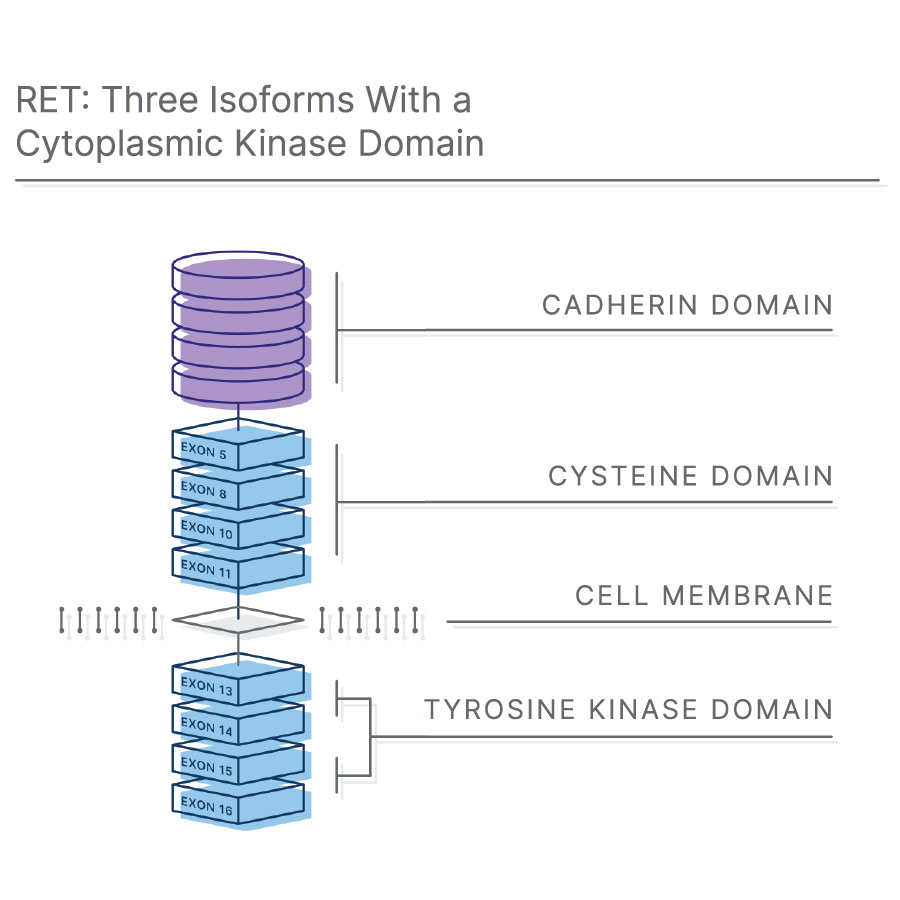
RET融合遺伝子とは?
RET融合は、複数のがんの病因に関与するがん遺伝子の活性化をもたらします。1-4。RETは、少なくとも12の個別遺伝子を融合パートナーとすることが知られており、KIF5B-RETは非小細胞肺がん(NSCLC)で最も頻繁に観察されるRET融合遺伝子です5,6。
RET(Rearranged during Transfection)遺伝子は、膜貫通型受容体チロシンキナーゼをコードしています7。RETは、グリア細胞株由来の神経栄養ファミリーリガンド(GFL)の受容体として機能します。GFLは、胚形成やヒトの発達において非常に重要な可溶性神経栄養因子のグループの一つです8,9。
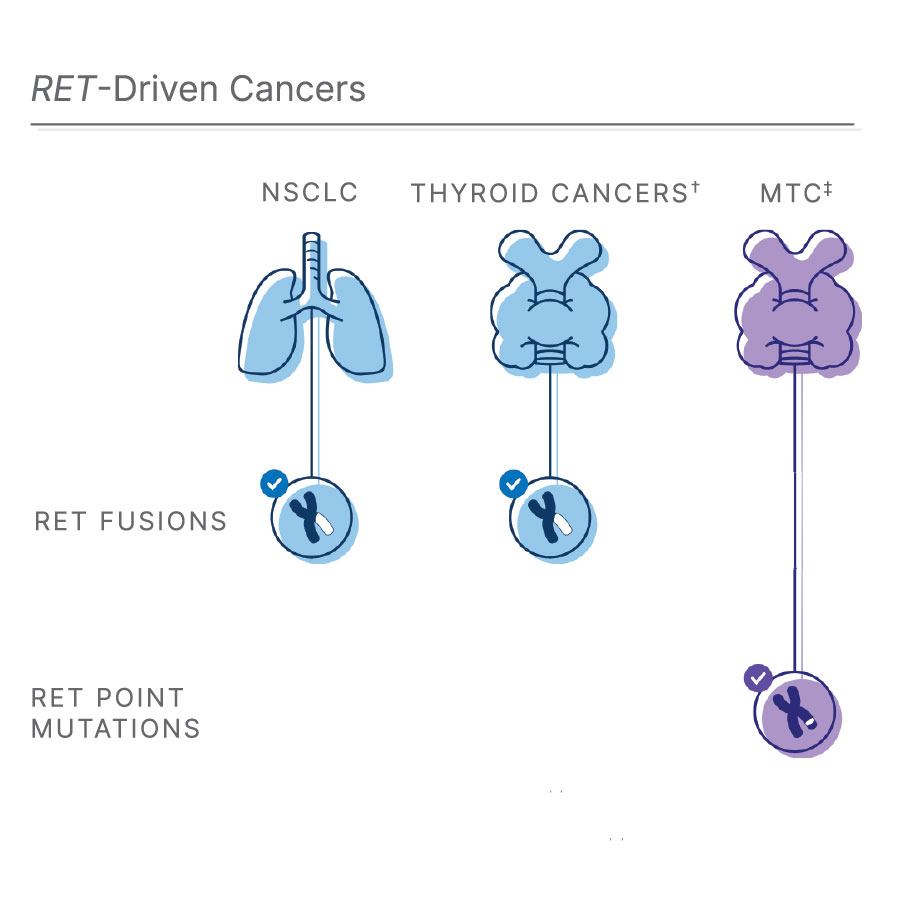
†RET融合遺伝子は甲状腺乳頭がん(PTC)の10~20%にも起こります1, 11, 12
‡甲状腺髄様がん(MTC):RET点変異はほとんどのMTCに影響を及ぼします1, 3, 12
RET融合遺伝子は最も一般的なタイプの肺がんで成長促進因子として関与している
毎年200万人以上が肺がんを罹患し、その症例の85%は非小細胞肺がん(NSCLC)です10。NSCLCに罹患している患者のうち、約2%はRET融合遺伝子が関与しています1,8。また、RET融合遺伝子は甲状腺乳頭がん(PTC)の10~20%にも発生しています1,11,12。
RET変異は稀ですが、RET阻害薬によって高度にアクショナブルな変異となります。選択的RET阻害剤RETEVMO®(selpercatinib)およびGAVRETO®(pralsetinib)のFDA承認を受け、RETは転移性NSCLCおよび甲状腺がん患者の検査になくてはならないバイオマーカーとなっています。さらに、RETEVMO®(selpercatinib)に腫瘍を横断した承認が得られたことにより、RET融合遺伝子の検出による進行性または転移性固形腫瘍患者への治療オプションが拡大されました。そのため、革新的プレシジョンメディシンを適用するには、あらゆるアクショナブルバイオマーカーの検査を実施する必要があります。
包括的ゲノムプロファイリングによる希少バイオマーカーの検出
例示した試験では、10,000例の患者のうちの37%にアクショナブル変異がCGPによって特定されました14。RET融合遺伝子に対する標的治療が利用可能であることを考察すると、NSCLCの分子特性評価の必要性はますます拡大しています。
DNA + RNAワークフローを使用すると、CGPは増加している一般的および希少ドライバー変異の評価を1回の検査で同時に実施することができます。すべてのバイオマーカーを一度に評価することで、アクショナブル変異を特定する機会を増やすことができます。
RET融合遺伝子の詳細について学ぶ

NSCLCにおけるRET変異の検査アプローチ
NSCLCに関わるバイオマーカーの展望は進化を続けており、新しい治療法はRET変異腫瘍をターゲットにしていることからRET変異検査の重要性はますます大きくなっています。このウェビナーで、Jaclyn Hechtman, MDによる解説をご視聴ください。

RET融合遺伝子の標的治療
新規高度選択的RET標的薬はRETを原因とするNSCLCを対象に試験され、標的治療を前進させています。このインフォグラフィックで、これらの発見を深く掘り下げてみましょう。

患者の成功事例:ステージIVの肺がんに打ち勝つ
AJ Patel氏の余命は6カ月でした。8年後に同氏は、バイオマーカー検査によってすべてが変化したと語ってくれました。
参考文献
- Kato S, Subbiah V, Marchlik E, Elkin SK, Carter JL, Kurzrock R. RET Aberrations in Diverse Cancers: Next-Generation Sequencing of 4,871 Patients. Clin Cancer Res. 2017;23(8):1988-1997. doi:10.1158/1078-0432.CCR-16-1679
- Drilon A, Hu ZI, Lai GGY, Tan DSW. Targeting RET-driven cancers: lessons from evolving preclinical and clinical landscapes. Nat Rev Clin Oncol. 2018;15(3):150. doi:10.1038/nrclinonc.2017.188
- Pietrantonio F, Di Nicolantonio F, Schrock AB, et al. RET fusions in a small subset of advanced colorectal cancers at risk of being neglected. Ann Oncol. 2018;29(6):1394-1401. doi:10.1093/annonc/mdy090
- Su X, He C, Ma J, et al. RET/PTC Rearrangements Are Associated with Elevated Postoperative TSH Levels and Multifocal Lesions in Papillary Thyroid Cancer without Concomitant Thyroid Benign Disease. PLoS One. 2016;11(11):e0165596. 発行日:2016年11月1日。doi:10.1371/journal.pone.0165596
- Takeuchi K, Soda M, Togashi Y, et al. RET, ROS1 and ALK fusions in lung cancer. Nat Med. 2012;18(3):378-381. 発行日:2012年2月12日。doi:10.1038/nm.2658
- Lipson D, Capelletti M, Yelensky R, et al. Identification of new ALK and RET gene fusions from colorectal and lung cancer biopsies. Nat Med. 2012;18(3):382-384. 発行日:2012年2月12日。doi:10.1038/nm.2673
- Ibáñez CF. Structure and physiology of the RET receptor tyrosine kinase. Cold Spring Harb Perspect Biol. 2013;5(2):a009134. doi:10.1101/cshperspect.a009134
- Wang X. Structural studies of GDNF family ligands with their receptors-Insights into ligand recognition and activation of receptor tyrosine kinase RET. Biochem Biophys Acta. 2013;1834:2205-2212
- de Graaff E, Srinivas S, Kilkenny C, et al. Differential activities of the RET tyrosine kinase receptor isoforms during mammalian embryogenesis. Genes Dev. 2001;15(18):2433‐2444. doi:10.1101/gad.205001
- International Agency on Cancer. World Health Organization. Global Cancer Observatory website. アクセス日:2020年6月24日。https://gco.iarc.fr/
- Mulligan LM. RET revisited: expanding the oncogenic portfolio. Nat Rev Cancer. 2014;14(3):173-186. doi:10.1038/nrc3680
- Li AY, McCusker MG, Russo A, et al. RET fusions in solid tumors. Cancer Treat Rev. 2019;81:101911. doi:10.1016/j.ctrv.2019.101911
- Gautschi, O, Milia J, Filleron T, et al. Targeting RET in patients with RET-rearranged lung cancers: results from the global, multicenter RET registry. J Clin Oncol. 2017;35:1403-1410. doi:10.1200/JCO.2016.70.9352
- Zehir A, Benayed R, Shah RH, et al. Mutational landscape of metastatic cancer revealed from prospective clinical sequencing of 10,000 patients [published correction appears in Nat Med. 2017 Aug 4;23 (8):1004]. Nat Med. 2017;23(6):703-713. doi:10.1038/nm.4333